使用Python對Dicom文件進行讀取與寫入的實現
Pydicom
單張影像的讀取
使用 pydicom.dcmread() 函數進行單張影像的讀取,返回一個pydicom.dataset.FileDataset對象.
import osimport pydicom# 調用本地的 dicom file folder_path = r'D:FilesDataMaterials'file_name = 'PA1_0001.dcm'file_path = os.path.join(folder_path,file_name)ds = pydicom.dcmread(file_path)
在一些特殊情況下,比如直接讀取從醫院拿到的數據(未經任何處理)時,可能會發生以下報錯:
raise InvalidDicomError('File is missing DICOM File Meta Information 'pydicom.errors.InvalidDicomError: File is missing DICOM File Meta Information header or the ’DICM’ prefix is missing from the header. Use force=True to force reading.
可以看到,由于缺失文件元信息頭,無法直接讀取,只能強行讀取.這種情況可以直接根據提示,調整命令為:
ds = pydicom.dcmread(file_path,force=True)
但后續還會碰到:
AttributeError: ’Dataset’ object has no attribute ’TransferSyntaxUID’
在網上檢索后發現,可以通過設置TransferSyntaxUID來解決問題:
ds.file_meta.TransferSyntaxUID = pydicom.uid.ImplicitVRLittleEndian
這樣就大功告成了(這里實際上就提前接觸到了下面讀取Dicom Tags的內容了)
一些簡單處理
讀取成功后,我們可以對 Dicom文件 進行一些簡單的處理
讀取并編輯Dicom Tags
可以通過兩種方法來讀取Tag的值
使用的Tag的Description
print(ds.PatientID,ds.StudyDate,ds.Modality)
使用 ds.get() 函數. 函數內參數采用的是Tag ID.幾種簡單的打開Dicom文件的軟件(如RadiAnt DICOM Viewer)都可以直接看到.這里不再贅述.
ds.get(0x00100020) # 這里得到的是PatientID
讀取到相應的Tag值后, 也可以將其他的值寫入這些Tag.只要最后保存一下就可以了.
借助Numpy與PIL.Image
讀取Dicom文件后,可以借助Numpy以及圖像處理庫(如PIL.Image)來進行簡單的處理.
借助Numpy
import numpy as npdata = np.array(ds.pixel_array)
注意這里使用的是 np.array() 而不是 np.asarray(). 因為前者的更改并不會帶來原pixel_array的改變.在轉化為ndarray后 可以直接進行簡單的切割和連接,比如截取某一部分和將兩張圖像拼在一起等,之后再寫入并保存下來即可.
借助PIL.Image
from PIL import Imagedata_img = Image.fromarray(ds.pixel_array)data_img_rotated = data_img.rotate(angle=45,resample=Image.BICUBIC,fillcolor=data_img.getpixel((0,0)))
這里展示的是旋轉, 還有其他功能如resize等.需要注意的是,從Numpy的ndarray轉化為Image時,一般會發生變化:
print(data.dtype) # int16data_rotated = np.array(data_img_rotated)print(data_img) # int32
只需要指定參數就可以解決了
data_rotated = np.array(data_img_rotated,dtype = np.int16)
可視化
簡單的可視化Pydicom沒有直接的實現方法,我們可以通過上面借助Matplotlib以及Image模塊來實現.但效果有限.
借助 Matplotlib (Pydicom官方文檔中使用的方法)
from matplotlib import pyplotpyplot.imshow(ds.pixel_array,cmap=pyplot.cm.bone)pyplot.show()
效果如圖所示:
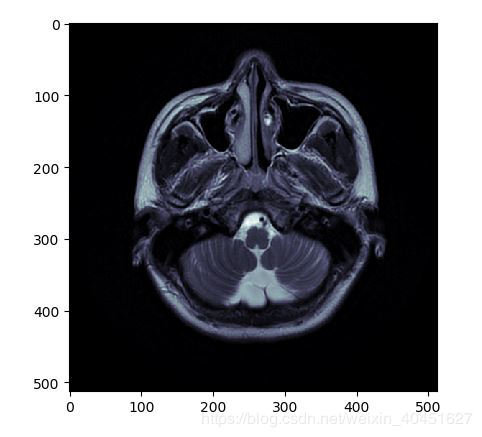
但真實的圖像是:
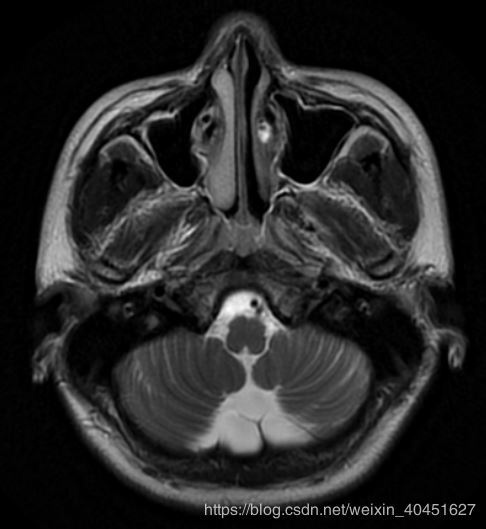
顯然顏色是有區別的.導致這種差別的原因是pyplot函數使用的cm也就是'color map' 是簡單的'bone' 并不能滿足醫學圖像的要求.
借助Image模塊
data_img.show()
一條指令即可,但是效果很差,如圖所示:
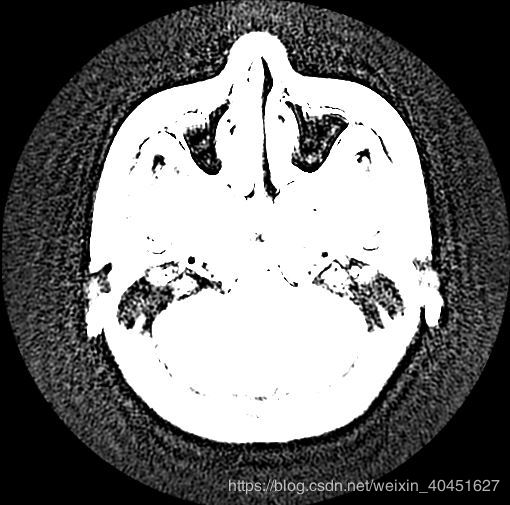
綜合來看,兩種方法都不是很好.
單張影像的寫入
經過上面對Tag值的修改, 對圖像的切割, 旋轉等操作.最后需要重新寫入該Dicom文件.
ds.PixelData = data_rotated.tobytes()ds.Rows,ds.Columns = data_rotated.shapenew_name = 'dicom_rotated.dcm'ds.save_as(os.path.join(folder_path,new_name))
SimpleITK
SimpleITK 是從基于C++的ITK遷移到Python的,所以很多方法的使用都跟C++很相似.
import SimpleITK as sitk
單張影像的讀取
有兩種方法:
sitk.ReadImage()這種方法直接返回image對象,簡單易懂.但是無法讀取Tag的值.
img = sitk.ReadImage(file_path)print(type(img)) # <class ’SimpleITK.SimpleITK.Image’>
sitk.ImageFileReader()這種方法比較像C++的操作風格,需要先初始化一個對象,然后設置一些參數,最后返回image.相對更復雜,但可以操作的點比較多
file_reader = sitk.ImageFileReader()file_reader.SetFileName(file_path) #這里只顯示了必需的,還有很多可以設置的參數data = file_reader.Execute()# 使用這種方法讀取Dicom的Tag Valuefor key in file_reader.GetMetaDataKeys(): print(key,file_reader.GetMetaData(key))
以上兩種方法返回的都是三維的對象,這與Pydicom有很大的不同.
data_np = sitk.GetArrayFromImage(data)print(data_np.shape) # (1, 512, 512) = (Slice index, Rows, Columns)
序列讀取
序列讀取的方法與單張圖像讀取的第二種方法很相似.(暫且只發現了一種方法讀取序列,如果還有其他方法,請在評論區予以補充,感謝!)
series_reader = sitk.ImageSeriesReader()fileNames = series_reader.GetGDCMSeriesFileNames(folder_name)series_reader.SetFileNames(fileNames)images = series_reader.Execute()
同樣,返回的也是三維的對象.
一些簡單操作
SimpleITK 包含很多圖像處理如濾波的工具,這里簡單介紹一個邊緣檢測工具和可視化工具
邊緣檢測
以Canny邊緣檢測算子為例,與讀取單張圖像類似,同樣有兩種方式:
sitk.CannyEdgeDetection()由于濾波的對象必須是32位圖像或者其他格式, 需要通過 sitk.Cast() 轉換. 之后可以再轉換回原格式.
data_32 = sitk.Cast(data,sitk.sitkFloat32)data_edge_1 = sitk.CannyEdgeDetection(data_32,5,30,[5]*3,[0.8]*3)
sitk.CannyEdgeDetectionImageFilter()這個操作相對麻煩一些
Canny = sitk.CannyEdgeDetectionImageFilter()Canny.SetLowerThreshold(5)Canny.SetUpperThreshold(30)Canny.SetVariance([5]*3)Canny.SetMaximumError([0.5]*3)data_edge_2 = Canny.Execute(data_32)
可視化
可視化的方法非常簡單 只需要一條指令:
sitk.Show()
但需要先安裝工具ImageJ,否則無法使用.具體的安裝鏈接,可以參考這篇博文:sitk.show()與imageJ結合使用常見的問題
同一張Dicom文件使用sitk.Show()得到的效果如下圖:
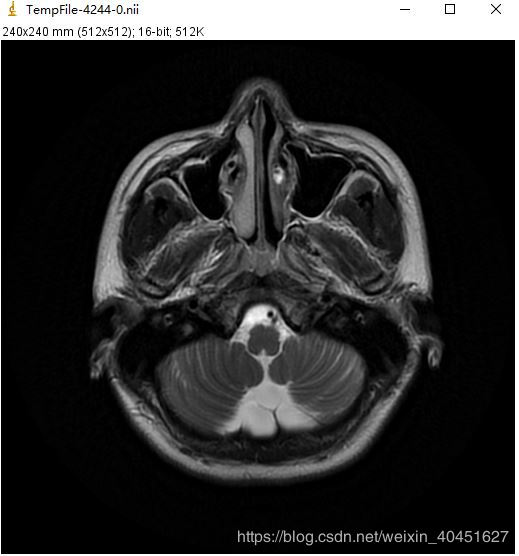
除此之外,ImageJ還有一個Tool Bar 支持對圖像的進一步處理:
可見,SimpleITK的可視化要比上面介紹的強大很多,不僅可以實現單張圖像的可視化以及圖像處理,還可以同時對整個序列的圖像進行統一處理.
單張影像的寫入
同樣有兩種方法
sitk.WriteImage()
new_name = 'new_MR_2.dcm'sitk.WriteImage(img,os.path.join(folder_name,new_name))
sitk.ImageFileWriter()
file_writer = sitk.ImageFileWriter()file_writer.SetFileName(os.path.join(folder_name,new_name))file_writer.SetImageIO(imageio='GDCMImageIO')file_writer.Execute(img)
使用這兩種方法進行寫入的時候,會發現,即便什么也沒有做,但得到的新Dicom文件要小于原始的Dicom文件.這是因為新的Dicom文件中沒有Private Creator信息(屬于Dicom Tag的內容).當然如果原始Dicom文件中本就沒有這種信息,文件大小是保持相同的.因為很多時候只是對圖像進行處理,所以不再深究.
到此這篇關于使用Python對Dicom文件進行讀取與寫入的實現的文章就介紹到這了,更多相關Python Dicom文件進行讀取與寫入內容請搜索好吧啦網以前的文章或繼續瀏覽下面的相關文章希望大家以后多多支持好吧啦網!
相關文章:
 網公網安備
網公網安備